lundi, 6 mars 2023
Journal de bord de notre cerveau à tous les niveaux : des cartes cérébrales à différentes échelles
.
Étant toujours dans la phase de relecture finale de mon livre jusqu’à la fin du printemps, je continue son « journal de bord » en y publiant certains encadrés qui n’ont pu, faute d’espace, trouver leur place dans le bouquin. Celui-ci entretenant déjà des rapports étroits avec le site web Le cerveau à tous les niveaux et son blogue grâce à différents renvois, cette conversion ne fait donc qu’étendre une approche déjà présente depuis le début du projet. Je poursuis donc aujourd’hui le « nettoyage » du chapitre 5 avec un encadré qui rappelle que lorsqu’on fait la cartographie d’un territoire le moindrement complexe, on fait face à un problème d’échelle. Dans le cas de la cartographie cérébrale, on a des cartes à l’échelle « micro » où l’on a fait des reconstitutions 3D de la manière précise dont s’agencent nos connexions synaptiques, mais qui ne nous permettent évidemment pas d’avoir une idée d’ensemble des grands faisceaux nerveux à l’échelle du cerveau entier. Même chose si on s’élève à un niveau « méso » où l’on a pu tracer le trajet complet d’axones de leur origine jusqu’à leur destination dans le cerveau de souris. On perd alors les détails des connexions fines de chaque neurone. Et à l’échelle « macro », où l’on a pu faire de fines tranches de cerveaux humains entiers et les numériser ensuite pour avoir une carte quand même très précise des moindres noyaux cérébraux, on ne peut pas voir le trajet des axones et encore moins l’emplacement des synapses. Bref, on ne peut jamais tout voir en même temps, mais chacune de ces échelles nous apportent séparément de précieuses informations sur la structure de notre cerveau.
* * *
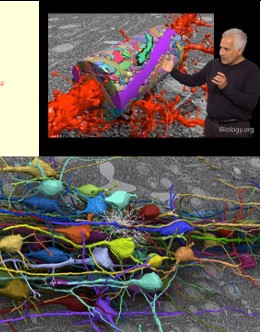
À la plus petite échelle, l’échelle micro, il faut utiliser la microscopie électronique capable d’atteindre le niveau de résolution de la synapse. Par exemple, avec le projet « EyeWire » que l’on pourrait traduire par « le câblage de l’œil », l’Américain Sebastian Seung s’est concentré uniquement sur un sous-groupe de neurones appelées « cellules ganglionnaires J » de la rétine de la souris.
C’est aussi la démarche de Jeff Lichtman, un autre Américain, avec sa fameuse coloration Brainbow (voir aussi ici et là) mais aussi avec une approche de reconstitution 3D à partir de coupes sérielles à l’échelle micro (voir aussi ici et là et là).
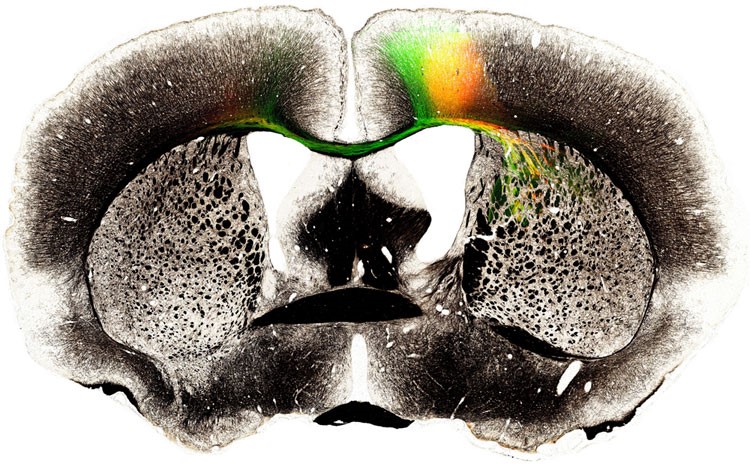
À l’échelle « meso », t’aurais par exemple le projet de cartographie « Mouse Brain Architecture Project » du cerveau entier de la souris à une échelle plus fine que celle que l’on peut obtenir avec l’imagerie cérébrale, mais allant moins dans le détail que la microscopie électronique capable de montrer les synapses.
Ou encore le « Mouse Connectome Project (MCP) », qui a utilisé des traceurs antérogrades mais aussi rétrogrades pour bien marquer les voies nerveuses dans les deux sens.
À partir de ce genre de cartes, on a lancé en 2018 le premier atlas virtuel en 3D de toutes les cellules du cerveau de la souris.
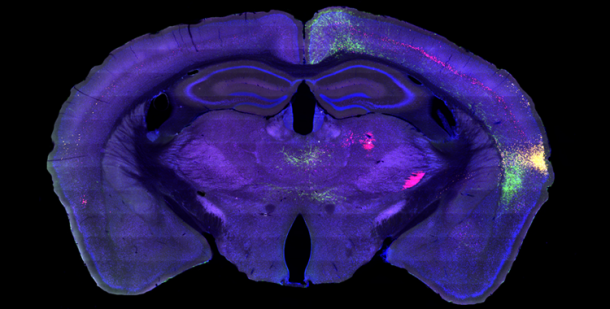
À l’échelle « macro » du cerveau humain maintenant, il y a différents projets comme celui du BigBrain de l’Institut neurologique de McGill et d’une équipe allemande qui, en 2013, ont tranché, imagée et analysé le cerveau d’une femme de 65 ans pour créer l’une des cartes 3D la plus détaillée de l’intégralité d’un cerveau humain. L’atlas a été réalisé grâce à la compilation de 7400 des tranches de ce cerveau conservé dans de la paraffine, chacune plus fine qu’un cheveu humain (20-microns). Il a fallu 1000 heures pour les imager à l’aide d’un scanner pour reconstruire le modèle 3D du cerveau sur un ordinateur. Des cerveaux de référence avaient déjà été cartographiés avec l’IRMf, mais ils n’ont une résolution que de 1 mm cube alors que les tranches de 20 µm de BigBrain permettent une résolution 50 fois meilleure. Ça ressemble à ce qu’on avait fait avec le cerveau du patient H.M. après son décès.
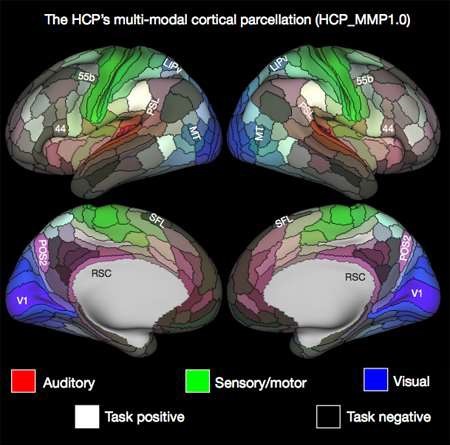
Finalement, une cartographie importante du cerveau entier réalisée grâce à plusieurs techniques d’imagerie cérébrale a été publiée en 2016 par Matthew Glasser, David Van Essen et leur équipe. En se basant sur des données du Human Connectome Project, ils ont pu caractériser 180 régions cérébrales par hémisphère, soit 97 nouvelles régions en plus des 83 déjà connues, délimitées par des changements nets dans la cytoarchitecture, la fonction, la connectivité et/ou la topographie (voir ici et là et là (p.130-136)).
Derrière tous ces travaux, il y a l’idée du « connectome », c’est-à-dire faire la cartographie anatomique la plus détaillée possible du système nerveux d’un organisme. Le seul animal dont on a le connectome complet est le ver C. elegans avec ses 302 neurones et ses 7000 synapses et il y a tout un débat à savoir si ça nous a réellement apporté quelque chose au niveau de la compréhension de son comportement. Pour ce qui est du connectome humain, c’est une carte qu’il sera toujours impossible à dresser d’une façon définitive à cause de la plasticité inhérente de ses connexions à l’échelle microscopique. Ce qui fait que la connectivité du cerveau d’un individu peut lui être spécifique, un peu comme une empreinte digitale, et en même temps se modifier au fil des décennies ou même varier significativement à l’échelle de la semaine ou du mois, comme l’a montré par exemple le psychologue Russell Poldrack qui a scanné son propre cerveau deux fois par semaine pendant un an et demi ! Mais un peu comme pour la cartographie du génome humain qui a été achevée en 2003 sans nous permettre de comprendre d’un coup toutes les maladies génétiques, de même on ne croit pas que la carte générale du connectome humain nous permettra de comprendre instantanément les maladies mentales. Comme pour le génome, elle pourra toutefois aider à élaborer de nouvelles hypothèses que l’on ne peut sans doute même pas imaginer aujourd’hui.
Non classé | Pas de commentaires