lundi, 17 août 2015
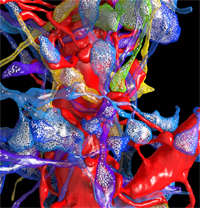
Une représentation 3D à l’échelle du nanomètre du cortex de souris
 En mai dernier, j’ai fait deux randonnées pédestres au mont Royal à Montréal en présentant celui-ci comme un modèle à grande échelle de la « forêt de neurones » que constitue notre cerveau. L’analogie n’était pas nouvelle, mais le fait d’observer les branches des arbres s’entrecroiser comme si c’était des axones et des dendrites donnait une petite « valeur ajoutée » à la métaphore. Dans la partie la plus dense de la forêt, on en venait même à se demander si voir chaque bourgeon qui s’ouvrait comme des épines dendritiques recevant autant de connexions des branches qui l’entouraient n’était pas un peu exagéré.
En mai dernier, j’ai fait deux randonnées pédestres au mont Royal à Montréal en présentant celui-ci comme un modèle à grande échelle de la « forêt de neurones » que constitue notre cerveau. L’analogie n’était pas nouvelle, mais le fait d’observer les branches des arbres s’entrecroiser comme si c’était des axones et des dendrites donnait une petite « valeur ajoutée » à la métaphore. Dans la partie la plus dense de la forêt, on en venait même à se demander si voir chaque bourgeon qui s’ouvrait comme des épines dendritiques recevant autant de connexions des branches qui l’entouraient n’était pas un peu exagéré.
À la lumière des dernières images produites par l’équipe de Jeff Lichtman et Narayanan Kasthuri (voir les deux premiers liens ci-dessous), on serait plutôt porté à se trouver un peu naïf tellement la complexité de la connectivité cérébrale dépasse, et de loin, celle de des arbres d’une forêt, aussi dense soit-elle !
Lichtman le répète depuis longtemps : c’est en allant à l’échelle de résolution du microscope électronique et en reconstituant tranche par tranche les moindres contacts entre les axones, les dendrites et les cellules gliales avoisinantes que nous pourrons voir apparaître des patterns qui nous échappent aux échelles plus « macros » utilisées jusqu’ici pour construire nos modèles de connectivité neuronale.
Il n’y a qu’à visionner le court vidéo de 45 secondes du premier lien ci-bas ou le vidéo du troisième pour constater d’abord qu’il n’y a pas de « vide » dans cortex cérébral de souris présenté ici (contrairement à l’espace entre les branches de la forêt). Tout l’espace est rempli de ces prolongements cellulaires qui se touchent et s’entrelacent inextricablement. Et c’est là que la reconstitution 3D par ordinateur montre toute sa puissance en pouvant faire apparaître ou disparaître à loisir tantôt seulement certains types d’axones (myélinisés ou pas, par exemple), tantôt certains types de dendrites, ou encore certaines cellules gliales et pas d’autres (astrocytes ou oligodendrocytes), etc.
Dans l’autre vidéo d’environ 5 minutes présentant la technique utilisée pour créer ces spectaculaires images en 3D, Lichtman rappelle qu’on a longtemps cru, en vertu de la règle dite « de Peter » (à ne pas confondre avec le principe d’incompétence du même nom…), que si l’on connaissait suffisamment la position d’un axone et d’un dendrite, on pouvait déduire les zones de contact formant les synapses entre les deux. Ce n’est malheureusement pas ce que montrent les images qu’ils ont obtenues. Celles-ci révèlent plutôt une irrégularité que seule une meilleure connaissance des propriétés moléculaires des axones et des épines dendritiques permettra d’élucider.
Comme souvent en science, on constate donc qu’une avancée importante repousse les questions à un niveau d’analyse qui devra être encore plus fin. Ça peut sembler décourageant, mais comme le dit Lichtman à la fin du vidéo : il n’y a jamais eu de cas, en biologie, ou en savoir plus était moins bon qu’en savoir moins. Ou encore, au risque de me répéter, cette fameuse pensée de Katz et Rosenzweig déjà citée dans le billet sur le rôle sous-estimé des péricytes, qui saisit si bien l’essence de la démarche scientifique :
« We have not succeeded in answering all our problems—indeed we sometimes feel we have not completely answered any of them.
The answers we have found have only served to raise a whole set of new questions.
In some ways we feel that we are as confused as ever, but we think we are confused on a higher level and about more important things.”
Tout ce que je peux donc espérer c’est que la lecture des travaux que je rapporte dans ce blogue vous laisse confus, mais à un niveau de confusion un peu plus élevé et pour des choses un peu plus importantes qu’avant leur lecture… 😉
![]() 3D brain map reveals connections between cells in nano-scale
3D brain map reveals connections between cells in nano-scale
![]() Saturated Reconstruction of a Volume of Neocortex
Saturated Reconstruction of a Volume of Neocortex
![]() Video: An incredibly detailed tour through the mouse brain
Video: An incredibly detailed tour through the mouse brain
Du simple au complexe | Comments Closed